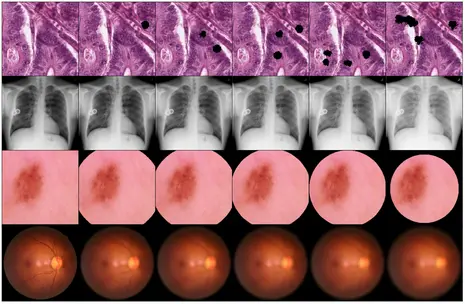
ADSMI-Workshop Paper: "MedMNIST-C: Comprehensive benchmark and improved classifier robustness by simulating realistic image corruptions"
Die Integration von neuronalen Netzwerken in die klinische Praxis wird durch Herausforderungen bei der Dom?nengeneralisierung und Robustheit eingeschr?nkt. Um diese zu adressieren, wurde MedMNIST-C, ein umfassender Benchmark-Datensatz, der auf der MedMNIST+ Sammlung basiert, eingeführt. MedMNIST-C deckt 12 Datens?tze und 9 Bildgebungsmodalit?ten ab und simuliert aufgaben- und modalit?tsspezifische Bildverzerrungen unterschiedlicher Schweregrade, um die Robustheit von Algorithmen gegenüber realen Artefakten und Verteilungsverschiebungen zu bewerten. Quantitative Beweise zeigen, dass einfach zu verwendende künstliche Verzerrungen eine hochleistungsf?hige, leichte Datenaugmentation erm?glichen, die die Robustheit des Modells verbessert. Im Gegensatz zu traditionellen, generischen Augmentierungsstrategien nutzt dieser Ansatz Dom?nenwissen und zeigt eine deutlich h?here Robustheit.
Für weitere Details k?nnen Sie das Paper hier und den zugeh?rige Programmcode hier abrufen.
Hauptkonferenz Paper: "Self-supervised Vision Transformer are Scalable Generative Models for Domain Generalization"
Trotz bemerkenswerter Fortschritte wurde die Integration von Deep-Learning-Techniken (DL) in klinisch relevante Anwendungen, insbesondere im Bereich der digitalen Histopathologie, durch Herausforderungen bei der robusten Generalisierung über verschiedene Bildgebungsdom?nen und -merkmale hinweg behindert. Traditionelle Milderungsstrategien in diesem Bereich, wie Datenaugmentation und Farbnormalisierung von F?rbungen, haben sich als unzureichend erwiesen, um dieses Problem zu l?sen, was die Erforschung alternativer Methoden erforderlich macht. Zu diesem Zweck wird eine neuartige generative Methode zur Dom?nengeneralisierung in histopathologischen Bildern vorgeschlagen. Diese Methode verwendet einen generativen, selbstüberwachten Vision Transformer, um dynamisch Merkmale von Bildausschnitten zu extrahieren und nahtlos in die Originalbilder einzufügen, wodurch neuartige synthetische Bilder mit vielf?ltigen Attributen entstehen. Durch die Anreicherung des Datensatzes mit solchen synthetisierten Bildern soll dessen ganzheitliche Natur verbessert werden, was die Generalisierung von DL-Modellen auf unbekannte Dom?nen erleichtert. Umfangreiche Experimente, die an zwei unterschiedlichen histopathologischen Datens?tzen durchgeführt wurden, zeigen die Wirksamkeit dieses Ansatzes und übertreffen den Stand der Technik deutlich, sowohl beim Camelyon17-wilds Challenge-Datensatz (+2%) als auch bei einem zweiten Epithel-Stroma-Datensatz (+26%). Darüber hinaus wird die F?higkeit dieser Methode betont, mit der zunehmenden Verfügbarkeit von nicht gekennzeichneten Datenproben und komplexeren, h?her parametrischen Architekturen leicht zu skalieren.
Für weitere Details k?nnen Sie das Paper hier und den zugeh?rige Programmcode hier abrufen.
?ber MICCAI und ADSMI
Die MICCAI-Konferenz wird als erstklassige j?hrliche Veranstaltung anerkannt, die sich auf die neuesten Entwicklungen in der medizinischen Bildverarbeitung, computerassistierten Intervention und verwandten Bereichen konzentriert. Sie zieht erstklassige Forschung aus aller Welt an, f?rdert Zusammenarbeit und Fortschritte in der medizinischen Bildgebung KI.
Der MICCAI-Workshop zur F?rderung von Datenl?sungen in der medizinischen Bildgebung KI (ADSMI) bietet eine Plattform für Diskussionen und Innovationen an der Schnittstelle von medizinischer Bildgebung, KI und Datenwissenschaft. Dieser Workshop befasst sich mit den Herausforderungen der Datenknappheit, -qualit?t und -interoperabilit?t in der medizinischen Bildgebung KI und vereint die Bemühungen von Data Augmentation, Labeling, and Imperfections, Big Task Small Data, und Medical Image Learning with Limited and Noisy Data.
Freuen Sie sich auf spannende Einblicke und Updates direkt von der MICCAI 2024 im Oktober!